Maximum Parsimony Analyse
Sinn und Zweck
Maximum Parsimony ist eine mögliche Methode einen Stammbaum zu generieren. Die wörtliche Übersetzung von "Maximum Parsimony" ins Deutsche ist "Maximale Sparsamkeit" was die Methode treffend beschreibt. Der evolutionäre Wandel, der jedem Stammbaum unterliegt, wird in dieser Methode als "realistisch" angesehen wenn er mit "maximal" wenigen Veränderungsannahmen auskommt.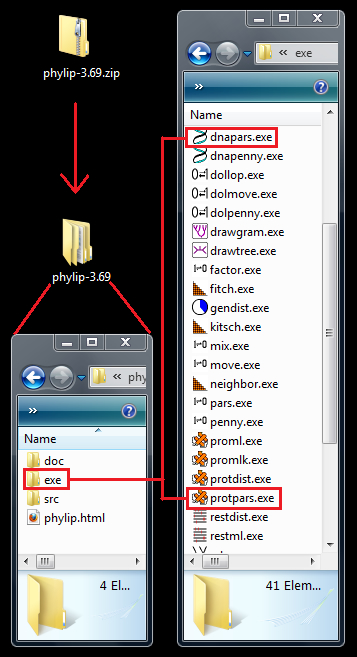
Erste Schritte ...
1. Das Programmpaket hier je nach Betriebssystem downloaden und extrahieren.
2. Die benötigten Programme z.B. in einen eigenen Arbeitsordner kopieren ("MP-Analyse")
3. Bei weiteren Fragen sei auf den doc-Ordner über dem exe-Ordner verwiesen
4. Daten im Phylip 4 (*phy) Format z.B. aus BioEdit ebenfalls in den Ordner kopieren
5. Je nach Datensatz ( DNA oder Protein ) das entsprechende Programm öffnen
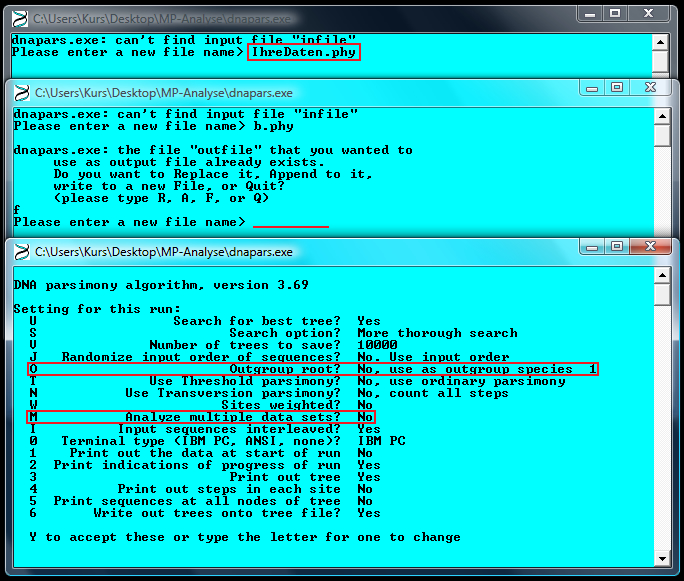
6. Hier beispielhaft der weitere Weg mit dnapars.exe und IhreDatei.phy
7. Nach dem Starten werden Sie zunächst dazu aufgefordert den Filename ( IhreDatei.phy ) anzugeben, mit Dateiendung.
8. Nach der Bestätigung mit "Enter" gelangen Sie zu den Settings, ist dies nicht Ihre erste Analyse werden Sie eventuell dazu aufgefordert einen neuen Namen für ihr 'Outfile' anzugeben.
9. Zu jede Option finden Sie eine ausführliche Erklärung im phylip.html File bei "main documentation file"
10. Wichtig für Sie sind anfangs (O) für Outgroup root und (M) für multiple data sets bei Daten die Sie mittels seqboot.exe erzeugt haben
11. Mit 'Y' und 'Enter' können Sie nun die Analyse starten; auch hier werden Sie eventuell dazu aufgefordert neue Filenamen anzugeben.
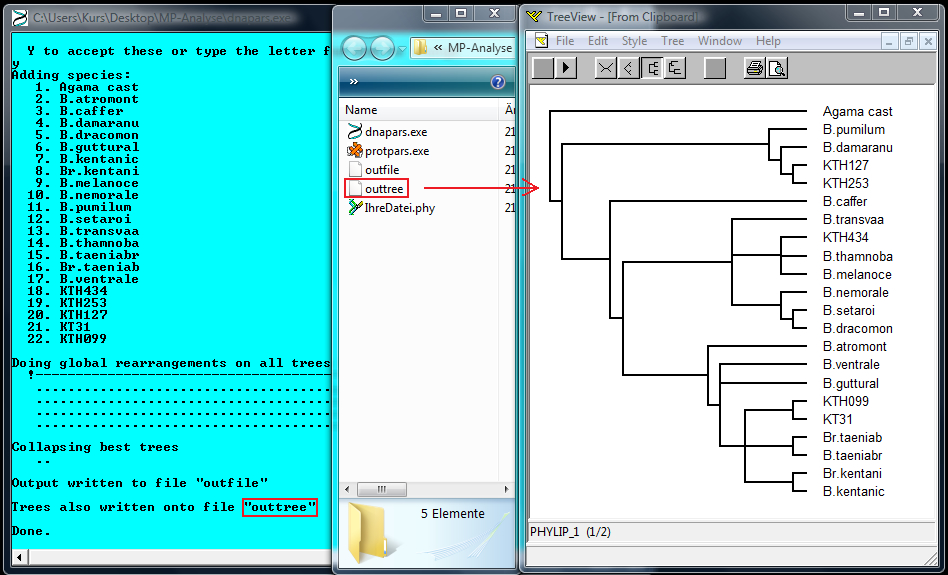
12. Final können Sie sich nun den errechneten Baum z.B. mit TreeView anzeigen lassen