Programme zur grafischen Darstellung von Stammbäumen

Treeview steht kostenlos für alle gänigen Betriebssysteme zum Download zur Verfügung.
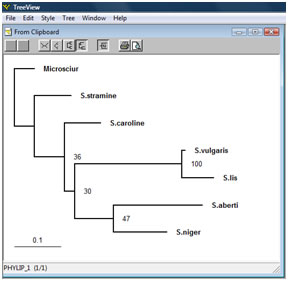
Treeview erstellt Stammbäume aus tree-files, die durch die Bearbeitung der Datensätze mit den oben genannten Programmen erzeugt wurden.
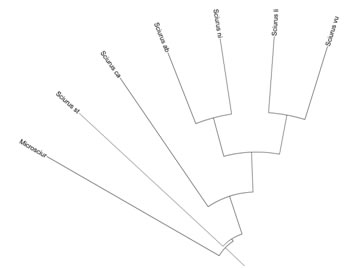
Die erstellten Stammbäume lassen sich in verschiedenen Formen anzeigen, z.B. als Phylogram oder ungegabelte Stammbäume.
Wenn ein Consensbaum erstellt wurde, lassen sich auch die Unterstützungswerte an den Knoten anzeigen, außerdem lassen sich die Stammbäume im nach hinein noch verändern.
Stammbäume können bis zu 100 Taxa enthalten.
Treeview überzeugt durch das einfache Handhabung
Link: iTOL - Interactive tree of life

iTOL ist ein Online-tool zum Erstellen und Bearbeiten von Stammbäumen.
Zum Hochladen der Datensätze müssen diese in einem dieser Formate vorliegen: Newick, Nexus oder PhyloXML.
Es besteht nicht nur die Möglichkeit eigene Datensätze hochzuladen, sondern man kann sich direkt Daten vom NCBI-Taxonomy-Browser über die IDs Daten laden..
Es können verschieden Daten am Baum angegeben werden, wie die Genomgröße und Proteindomänen.
Die erstellten Stammbäume können auch exportiert werden, in vielen verschiedenen Formaten.
Die Möglichkeiten der Bearbeitung der Stammbäume ist auf iTOL höher als bei Treeview, aber dafür muss man sich mit iTOL auch länger vertraut machen.